“전장유전체로 변이까지 잡는다”…서울대병원, 한국인 난청 유전자 지도 구축
사람의 전체 유전정보를 정밀하게 해독하는 전장유전체분석(WGS)이 난청의 숨겨진 유전적 원인을 밝혀내며, 맞춤형 정밀의료의 새 지평을 열고 있다. 서울대병원 연구팀이 발표한 이번 연구는 기존 기술로 규명하지 못했던 변이까지 찾아내며, 한국인을 위한 난청 유전자 지도를 최초로 완성했다. 업계는 이번 연구가 유전체 분석 기반 맞춤치료 경쟁의 분기점이 될 수 있다고 보고 있다.
서울대병원 소아이비인후과 이상연 교수, 임상유전체의학과 채종희·이승복 교수, 이노크라스 고준영 박사, 스탠포드대 박성열 박사로 구성된 공동연구팀은 서울대병원 이비인후과 및 인공와우센터에서 난청 환자 394가계(총 752명)를 대상으로 단계별 유전자 분석을 시행했다. 연구팀은 단일유전자 검사, 자체 타깃패널검사(TPS), 전장엑솜분석(WES), 마지막으로 전장유전체분석(WGS)을 순차적으로 적용, 감각신경성 난청(SNHL)의 유전적 원인을 체계적으로 추적했다.
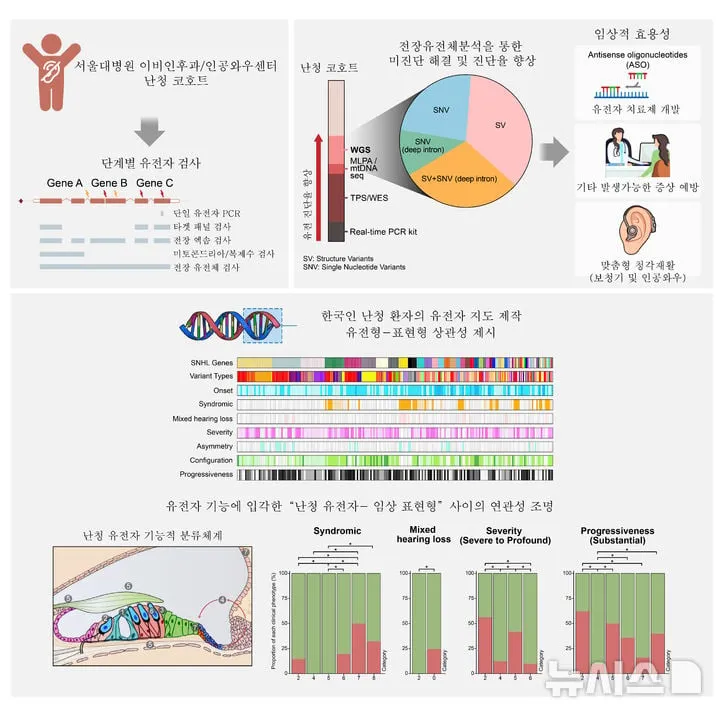
특히 전장유전체분석(WGS)은 기존 TPS·WES 등 검사 방식으론 발견할 수 없었던 ‘딥인트론 변이’(비코딩 영역 변이)와 유전자 내 ‘구조적 변이’를 포착해 진단율을 기존보다 19.2%(44가계) 높였다. 전체적으로 394가계 중 219가계에서 정확한 원인을 밝혀, 진단율이 사실상 20% 향상됐다. 딥인트론 변이란, 단백질 생성에 직접 관여하지 않는 유전자 비코딩 영역에서 발생하는 변이로, 새로운 RNA 스플라이싱(엑손·인트론 연결 과정) 장애와 연관돼 유전성 난청의 숨은 원인으로 주목받고 있다.
연구팀은 특히, 대표적 난청 유전자 USH2A에서 3종의 신규 딥인트론 변이를 확인했다. 이들 변이는 RNA 스플라이싱 오류를 유발, 단백질 생성에 장애를 일으키며, 곧바로 유전자 치료제 개발 후보로도 제시될 수 있다. 글로벌 정밀진단 분야에서 딥인트론·구조적 변이 분석은 아직 걸음마 단계인 만큼, 한국형 난청 유전자 지도 구축은 주목할 만한 성과로 평가받는다.
이러한 진단 도구의 발전은 실제 임상에서 미진단 환자의 정밀 치료로 연결될 가능성이 크다고 전문가들은 말한다. 기존 검사로 진단되지 않았던 난청 원인 환자군에 대해 향후 RNA 치료제 등 맞춤형 치료법 연구가 가능해지기 때문이다.
글로벌 시장에서는 미국 NIH, 영국 NHS 등도 전장유전체 분석 기반 정밀의료 사업을 시작하며 암·희귀질환, 유전성 질환 분야의 대규모 환자 데이터 지도를 구축 중이다. 이번 서울대병원 연구는 구조적 변이와 딥인트론 변이 첫 진단 적용, 한국인 특화 유전자 변이 데이터 확보 측면에서 경쟁 우위를 확보했다는 평가다.
다만 정밀유전자 분석 결과를 임상에 연계하려면, 안전성과 윤리기준에 근거한 추가 제도 정비 및 건강보험 인프라 확충 등 정책적 지원이 중요하다는 지적도 나온다. 데이터 보호와 AI 기반 해석 시스템의 신뢰성 강화, 규제기관(식약처 등)의 인증 절차 역시 본격 논의가 필요할 것으로 보인다.
이상연 서울대병원 교수는 “전장유전체분석으로 미진단 난청 환자의 정확한 유전적 원인 규명이 가능해졌다”며 “앞으로도 난청 미진단 원인에 대한 정밀한 분석, 맞춤형 유전자 치료 연결을 확대하는 데 집중할 계획”이라고 밝혔다. 전문가들은 “유전체 기반 정밀 진단은 향후 난청은 물론, 희귀·유전질환 치료 전반의 패러다임을 바꿀 기술”이라고 평가한다. 산업계는 전장유전체분석 기반 맞춤형 의료기술이 실제 임상현장에 안착할지 주시하고 있다.
